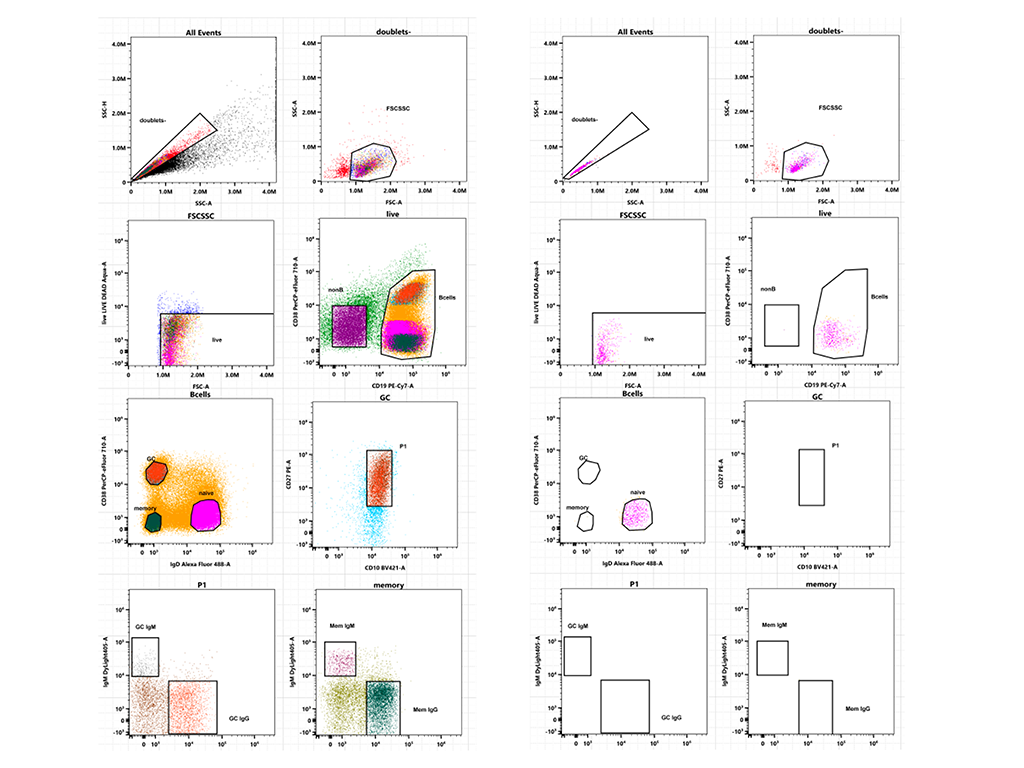
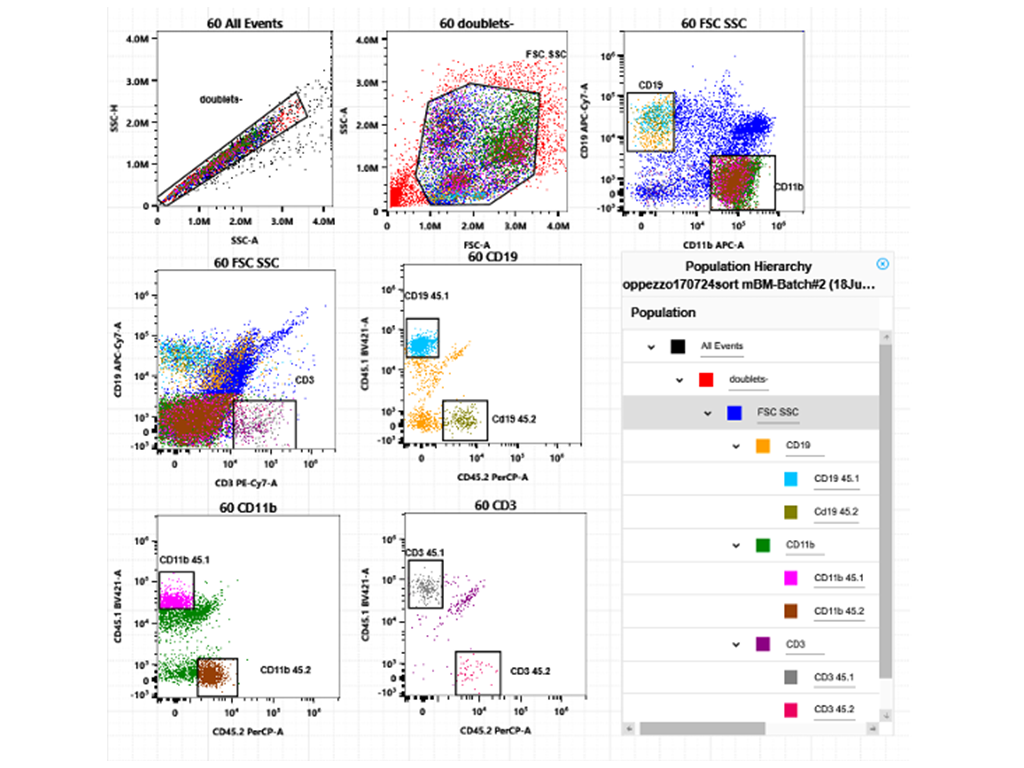
Citofluorimetria

Maria Grazia Totaro
Maria Grazia ha conseguito la laurea magistrale in Biologia applicata alla ricerca biomedica presso l’Università dell’Insubria a Varese. La sua formazione si è arricchita grazie a esperienze in istituti di ricerca di eccellenza come l’Istituto di Ricerca in Biomedicina (IRB) di Bellinzona e l’Istituto Europeo di Oncologia (IEO) e l’Istituto Humanitas di Milano, dove ha collaborato con ricercatori di spicco nel campo dell’immunologia. Durante il suo dottorato presso l’Istituto Humanitas, l’interesse per l’immunologia l’ha condotta a scoprire il mondo della citofluorimetria, rivelandone le straordinarie potenzialità. Conseguito il PhD, ha messo a frutto le competenze acquisite entrando a far parte dell’unità di Imaging di IFOM, sotto la guida del Dr. Dario Parazzoli, come tecnico specializzato in citofluorimetria. Nel corso di 10 anni, Maria Grazia ha consolidato le sue competenze e fornito un contributo significativo alla crescita e allo sviluppo delle attività di citofluorimetria dell’unità, diventandone un punto di riferimento. A ottobre 2024, è stata nominata Head of Facility dell’unità di Citofluorimetria, istituita a seguito della divisione dell’unità di Imaging, assumendo la responsabilità di guidare questa nuova realtà verso l’eccellenza scientifica.
Gallery Flow Cytometry unit
Facility members
Reference PI:
Massimiliano Pagani
Unit info
Sin dalla sua nascita negli anni '60, la citofluorimetria è stata uno strumento prezioso nella ricerca scientifica, offrendo una vasta gamma di applicazioni, acquisizione rapida dei dati e la possibilità di esplorare numerosi aspetti biologici. Negli ultimi anni, questa tecnologia ha vissuto un' evoluzione esponenziale. Mentre per molti anni il numero di parametri che gli strumenti potevano elaborare è rimasto relativamente basso, aumentando gradualmente nel tempo, l'applicazione della tecnologia full spectrum nella citometria a flusso ha consentito agli strumenti di analizzare più di 50 parametri contemporaneamente (come dimostrato e pubblicato). Inoltre, la capacità di separare le popolazioni cellulari riconosciute rende i citofluorimetri degli strumenti indispensabili nello studio soprattutto dell'immunologia, nonché nell'esplorazione di varie funzioni e meccanismi cellulari. La facility di citofluorimetria dell'IFOM offre tecnologie all'avanguardia e strumenti di ultima generazione, gestiti da un personale altamente qualificato con decenni di esperienza. Le applicazioni supportate includono: immunofenotipizzazione, analisi del ciclo cellulare, studi sul metabolismo, apoptosi e proliferazione cellulare, analisi delle proteine fluorescenti e rilevamento di citochine, chemiokine e proteine intra/extra-cellulari. Gli utenti della struttura hanno accesso sia ai citofluorimetri tradizionali (FACSCanto II, Attune NxT) che agli strumenti spettrali (Aurora 5L). L'uso indipendente degli strumenti è consentito solo dopo aver completato un corso teorico e pratico organizzato dal personale della facility. Per tutti gli strumenti di sorting (convenzionali: MoFlo Astrios e FACSAria IIU, e spettrali: Aurora CS), l'accesso è consentito solo con assistenza. La facility offre anche un servizio di analisi in collaborazione con il team di bioinformatica per analisi complesse ad alto numero di parametri. Inoltre, il personale della facility è disponibile per offrire consulenza e supporto prima e durante la progettazione degli esperimenti, la configurazione dei pannelli e per la risoluzione dei problemi.
Applications/Services
Servizi
La facility si occupa della configurazione, ottimizzazione e manutenzione degli strumenti.
Il personale della facility offre assistenza e consulenza per la preparazione dei campioni, la progettazione dei pannelli e la strategia di gating.
Inoltre, la facility di FC stabilisce contatti con le aziende per conoscere e testare le tecnologie e i reagenti all'avanguardia nel campo della citofluorimetria e mantiene contatti con altre core facilities per discutere e affrontare opportunità, problematiche e novità.
Applicazioni
Identificazione delle sottopopolazioni cellulari da diversi campioni e tessuti umani e murini tramite colorazione multiparametrica:
- Subpopolazioni immunitarie (Treg, Tnaive, Tconv, GC Bcell, Bcell naive, TIL, TAM…)
- Cellule staminali tumorali
- Cellule endoteliali
- Cellule di mieloma
- Fibroblasti cardiaci
Identificazione delle sottopopolazioni cellulari da sferoidi, organoidi, coculture
Analisi del ciclo cellulare e proliferazione cellulare in cellule di mammifero e lievito di PI, Sytox, BrdU, EdU, FUCCI, Cicline, Ki67
Analisi del metabolismo cellulare:
- Assorbimento del glucosio (2NDBG)
- Visualizzazione dei lipidi (Bodipy) e delle lipid droplets (Nile Red)
- Funzionamento dei mitocondri (JC1, Mitotracker, Mitosox)
- Rilevamento di proteine intracellulari (TMR HaloTag)
- Rilevamento di specie reattive dell'ossigeno (CellRox, DFCDA)
- Monitoraggio del rapporto ATP/ADP (sensore Perceval)
Analisi della morte cellulare/apoptosi: Caspase3, AnnV\PI, DRAQ7
Analisi di linee cellulari o cellule primarie(umane o murine)ingegnerizzate con CRISPR/Cas9 o transdotte e trasfettate con proteine fluorescenti come GFP, RFP, mCherry, Scarlet, Tomato, YFP…
Lineage tracking con marcatura RGB barcoding
Analisi dell'interazione proteica: kit Duolink flowPLA®
Analisi dell'endocitosi cellulare: assorbimento di destrano
Analisi dell'assorbimento di microplastiche in diverse linee cellulari e in tessuti murini
Quantificazione di antigen bound per cell (ABC) tramite QuantiBrite™ beads
Analisi dei dati con diversi software:
- Kaluza
- FlowJo
- FCSExpress
- SpectroFlo
- FACSDiva
Sorting di tutte le cellule/popolazioni sopra indicate:
- Sorting bulk
- Sorting su supporti speciali per successive analisi (vetrini per imaging, piastre da 96 wells per screening/sequenziamento/spettrometria di massa/high content imaging)
- Sorting di singola cellula in piastre da 96 o 384 wells
- Sorting con precauzioni particolari per analisi metabolomiche/analisi NGS
Main users of the facility (IFOM Research Groups):
Equipment
Publications
PMID: 37713487
Clonal cooperation through soluble metabolite exchange facilitates metastatic outgrowth by modulating Allee effect.
Hershey BJ, Barozzi S, Orsenigo F, Pompei S, Iannelli F, Kamrad S, Matafora V, Pisati F, Calabrese L, Fragale G, Salvadori G, Martini E, Totaro MG, Magni S, Guan R, Parazzoli D, Maiuri P, Bachi A, Patil KR, Cosentino Lagomarsino M, Havas KM. Sci Adv. 2023 Sep 15;9(37):eadh4184. doi: 10.1126/sciadv.adh4184. Epub 2023 Sep 15. Free PMC article.
PMID: 37108076
Short-Term Exposure to Benzo(a)Pyrene Causes Disruption of GnRH Network in Zebrafish Embryos.
Gentile I, Vezzoli V, Martone S, Totaro MG, Bonomi M, Persani L, Marelli F. Int J Mol Sci. 2023 Apr 7;24(8):6913. doi: 10.3390/ijms24086913. Free PMC article.
PMID: 35918044
HDAC6 inhibition decreases leukemic stem cell expansion driven by Hedgehog hyperactivation by restoring primary ciliogenesis.
Pezzotta A, Gentile I, Genovese D, Totaro MG, Battaglia C, Leung AY, Fumagalli M, Parma M, Cazzaniga G, Fazio G, Alcalay M, Marozzi A, Pistocchi A. Pharmacol Res. 2022 Sep;183:106378. doi: 10.1016/j.phrs.2022.106378. Epub 2022 Jul 30.
PMID: 33653693
CD4 T Cell-Dependent Rejection of Beta-2 Microglobulin Null Mismatch Repair-Deficient Tumors.
Germano G, Lu S, Rospo G, Lamba S, Rousseau B, Fanelli S, Stenech D, Le DT, Hays J, Totaro MG, Amodio V, Chilà R, Mondino A, Diaz LA Jr, Di Nicolantonio F, Bardelli A. Cancer Discov. 2021 Jul;11(7):1844-1859. doi: 10.1158/2159-8290.CD-20-0987. Epub 2021 Mar 2.
PMID: 33436662
MITO-Luc/GFP zebrafish model to assess spatial and temporal evolution of cell proliferation in vivo.
de Latouliere L, Manni I, Ferrari L, Pisati F, Totaro MG, Gurtner A, Marra E, Pacello L, Pozzoli O, Aurisicchio L, Capogrossi MC, Deflorian G, Piaggio G. Sci Rep. 2021 Jan 12;11(1):671. PMID: 33436662 Free PMC article.
PMID: 33015043
HDAC8: A Promising Therapeutic Target for Acute Myeloid Leukemia.
Spreafico M, Gruszka AM, Valli D, Mazzola M, Deflorian G, Quintè A, Totaro MG, Battaglia C, Alcalay M, Marozzi A, Pistocchi A. Front Cell Dev Biol. 2020 Sep 4;8:844. doi: 10.3389/fcell.2020.00844. eCollection 2020. PMID: 33015043 Free PMC article.
PMID: 33903766
Heme catabolism by tumor-associated macrophages controls metastasis formation.
Consonni FM, Bleve A, Totaro MG, Storto M, Kunderfranco P, Termanini A, Pasqualini F, Alì C, Pandolfo C, Sgambelluri F, Grazia G, Santinami M, Maurichi A, Milione M, Erreni M, Doni A, Fabbri M, Gribaldo L, Rulli E, Soares MP, Torri V, Mortarini R, Anichini A, Sica A. Nat Immunol. 2021 May;22(5):595-606. doi: 10.1038/s41590-021-00921-5. Epub 2021 Apr 26.
PMID: 32265223
Tumor-Derived Prostaglandin E2 Promotes p50 NF-κB-Dependent Differentiation of Monocytic MDSCs.
Porta C, Consonni FM, Morlacchi S, Sangaletti S, Bleve A, Totaro MG, Larghi P, Rimoldi M, Tripodo C, Strauss L, Banfi S, Storto M, Pressiani T, Rimassa L, Tartari S, Ippolito A, Doni A, Soldà G, Duga S, Piccolo V, Ostuni R, Natoli G, Bronte V, Balzac F, Turco E, Hirsch E, Colombo MP, Sica A. Cancer Res. 2020 Jul 1;80(13):2874-2888. doi: 10.1158/0008-5472.CAN-19-2843. Epub 2020 Apr 7.
PMID: 18467122
Macrophage polarization in tumour progression.
Sica A, Larghi P, Mancino A, Rubino L, Porta C, Totaro MG, Rimoldi M, Biswas SK, Allavena P, Mantovani A. Semin Cancer Biol. 2008 Oct;18(5):349-55. doi: 10.1016/j.semcancer.2008.03.004. Epub 2008 Mar 26. Review.
PMID: 21995571
Macrophages in cancer and infectious diseases: the 'good' and the 'bad'.
Porta C, Riboldi E, Totaro MG, Strauss L, Sica A, Mantovani A. Immunotherapy. 2011 Oct;3(10):1185-202. doi: 10.2217/imt.11.116. Review.
PMID: 26267538
RORC1 Regulates Tumor-Promoting "Emergency" Granulo-Monocytopoiesis.
Strauss L, Sangaletti S, Consonni FM, Szebeni G, Morlacchi S, Totaro MG, Porta C, Anselmo A, Tartari S, Doni A, Zitelli F, Tripodo C, Colombo MP, Sica A. Cancer Cell. 2015 Aug 10;28(2):253-69. doi: 10.1016/j.ccell.2015.07.006. Free article.
PMID: 19616341
Cellular and molecular pathways linking inflammation and cancer.
Porta C, Larghi P, Rimoldi M, Totaro MG, Allavena P, Mantovani A, Sica A. Immunobiology. 2009;214(9-10):761-77. doi: 10.1016/j.imbio.2009.06.014. Epub 2009 Jul 17. Review.
PMID: 21948460
Origin and Functions of Tumor-Associated Myeloid Cells (TAMCs).
Sica A, Porta C, Morlacchi S, Banfi S, Strauss L, Rimoldi M, Totaro MG, Riboldi E. Cancer Microenviron. 2012 Aug;5(2):133-49. doi: 10.1007/s12307-011-0091-6. Epub 2011 Sep 24. Free PMC article.
PMID: 23049782
The p50 subunit of NF-κB orchestrates dendritic cell lifespan and activation of adaptive immunity.
Larghi P, Porta C, Riboldi E, Totaro MG, Carraro L, Orabona C, Sica A. PLoS One. 2012;7(9):e45279. doi: 10.1371/journal.pone.0045279. Epub 2012 Sep 25. Free PMC article.
PMID: 17825402
The histone H3 lysine-27 demethylase Jmjd3 links inflammation to inhibition of polycomb-mediated gene silencing.
De Santa F, Totaro MG, Prosperini E, Notarbartolo S, Testa G, Natoli G. Cell. 2007 Sep 21;130(6):1083-94. doi: 10.1016/j.cell.2007.08.019. Epub 2007 Sep 6. Free article.
PMID: 22127978
Enhanced interferon regulatory factor 3 binding to the interleukin-23p19 promoter correlates with enhanced interleukin-23 expression in systemic lupus erythematosus.
Smith S, Gabhann JN, Higgs R, Stacey K, Wahren-Herlenius M, Espinosa A, Totaro MG, Sica A, Ball E, Bell A, Johnston J, Browne P, O'Neill L, Kearns G, Jefferies CA. Arthritis Rheum. 2012 May;64(5):1601-9. doi: 10.1002/art.33494. Free article.
PMID: 18716661
The histone H3 lysine 27-specific demethylase Jmjd3 is required for neural commitment.
Burgold T, Spreafico F, De Santa F, Totaro MG, Prosperini E, Natoli G, Testa G. PLoS One. 2008 Aug 21;3(8):e3034. doi: 10.1371/journal.pone.0003034. Free PMC article.