Screening Genomico Funzionale

Giuseppina D’Alessandro
Nata nel 1988, Giuseppina D’Alessandro si laurea in Biotecnologie Mediche presso l'Università di Bari. Dopo aver completato gli studi nel 2012, si trasferisce a Milano, dove consegue un dottorato in Medicina Molecolare presso l'IFOM. La sua ricerca di dottorato, sotto la guida del Dr. Fabrizio d’Adda di Fagagna, si concentra sullo studio del ruolo degli RNA e degli ibridi RNA-DNA nelle rotture a doppio filamento del DNA, tra le lesioni più dannose per il DNA. Terminato il dottorato, Giuseppina riceve una borsa di studio internazionale tramite il programma iCARE-2, co-finanziato da AIRC e dall’Unione Europea nell'ambito delle Azioni Marie Skłodowska-Curie. Questo prestigioso riconoscimento la porta nel Regno Unito, dove lavora nel laboratorio del Prof. Sir Steve Jackson presso l'Università di Cambridge. Durante la sua esperienza post-dottorato al Gurdon Institute e successivamente al Cancer Research UK Cambridge Institute, amplia le sue conoscenze sulla risposta al danno del DNA, utilizzando tecnologie avanzate di editing genomico, tra cui CRISPR knock-out, inibizione e base editing. Dopo cinque anni nel Regno Unito, Giuseppina torna in Italia per istituire e dirigere l'Unità di Screening di Genomica Funzionale presso IFOM. In questo nuovo ruolo, applica le sue competenze nell'editing genomico e nelle tecnologie di screening CRISPR per supportare gli scienziati dell'IFOM e studiare i meccanismi di risposta ai danni del DNA su larga scala. Nel corso della sua carriera, Giuseppina ha concepito e guidato numerosi progetti e ha coordinato collaborazioni con ricercatori di fama internazionale. È inoltre revisore per riviste internazionali, tra cui Nature Communications, e contribuisce al servizio di preprint PreLights.
Gallery Functional genomics screening unit
Facility members
Mario Cinquanta
Marisa Aliprandi

Unit info
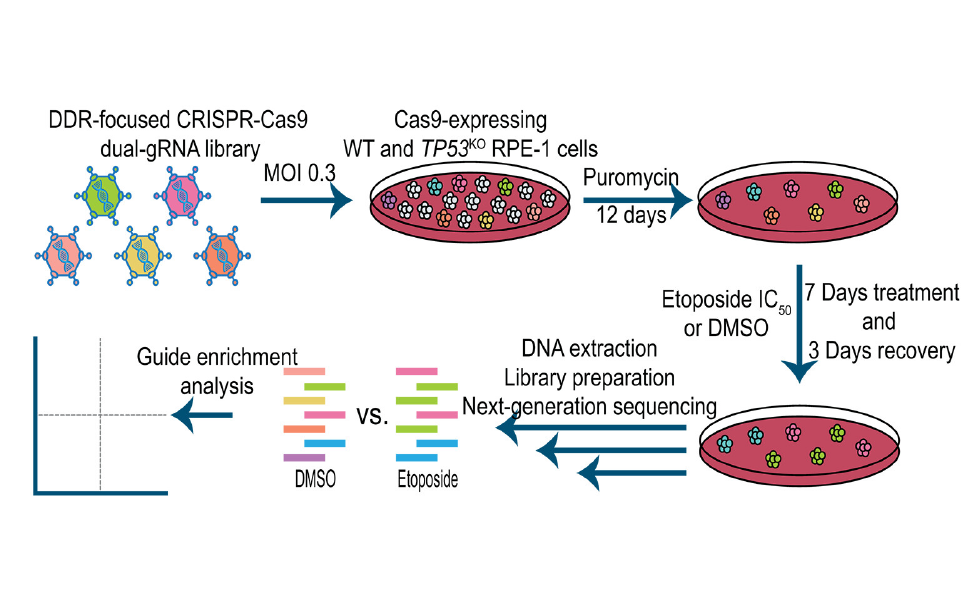
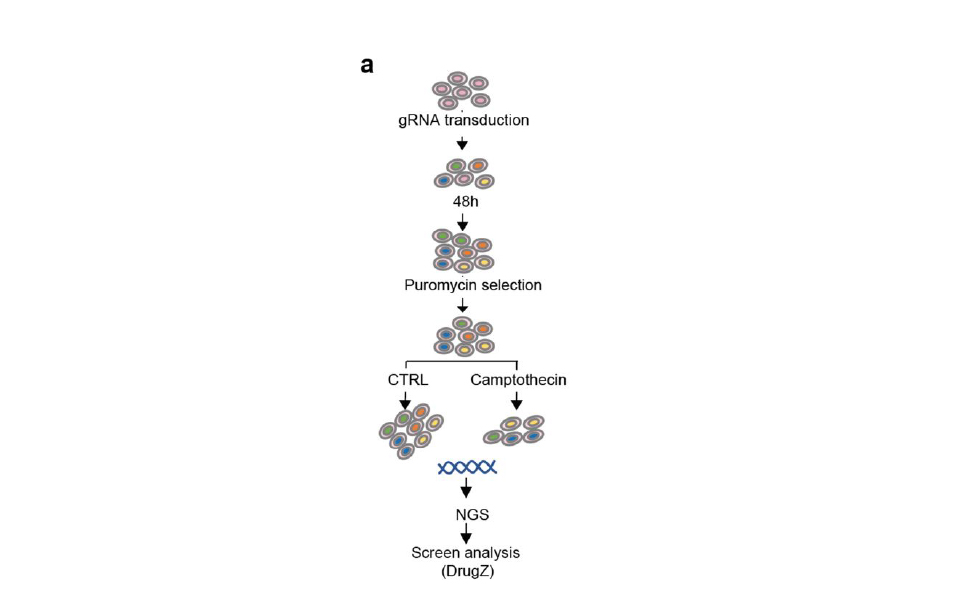
CRISPR/Cas9 è uno strumento rivoluzionario di editing genomico, derivato dai meccanismi di difesa naturali dei batteri. Quando infettati, i batteri incorporano segmenti di DNA virale nelle loro sequenze CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats). In caso di un nuovo attacco virale, l'enzima Cas9 (CRISPR-associated protein 9) utilizza le molecole di RNA trascritte dalle sequenze CRISPR per riconoscere il DNA virale e neutralizzare il virus. Nel 2012, le vincitrici del Premio Nobel per la Chimica Jennifer Doudna ed Emmanuelle Charpentier hanno adattato questo sistema alle cellule eucariotiche, creando uno strumento di editing genomico che taglia il DNA e sfrutta i meccanismi di riparazione cellulare per introdurre modifiche genetiche. L'Unità di Screening di Genomica Funzionale (FGS) conduce screening CRISPR su larga scala utilizzando librerie pooled o arrayed in vari modelli cellulari. Questa tecnologia consente di valutare gli effetti di migliaia di perturbazioni genetiche a livello della singola cellula attraverso approcci di CRISPR knockout (CRISPR KO), CRISPR interference (CRISPRi) e CRISPR activation (CRISPRa). Tale sistema utilizza inoltre base o prime editing per ricapitolare mutazioni specifiche, come quelle rilevanti per lo sviluppo e la progressione del cancro. Il base editing combina un modulo Cas con una deaminasi per convertire le basi del DNA, mentre il prime editing utilizza un templato di RNA per introdurre modifiche genetiche mirate nel DNA target. L'Unità FGS integra queste perturbazioni con vari readouts, tra cui vitalità cellulare e analisi fenotipiche complesse, spesso in collaborazione con Imaging, FACS Facility ed ETP. L'analisi dei dati è supportata dall'Unità di Bioinformatica IFOM. Collaborando con diversi gruppi IFOM, l'Unità FGS mira a chiarire le funzioni dei singoli geni, comprendere i loro ruoli nei meccanismi delle malattie, e ad identificare nuovi bersagli terapeutici. Oltre a supportare i ricercatori dell'IFOM sviluppando e ottimizzando strumenti basati su CRISPR/Cas, l'Unità adotta tali tecnologie per investigare la risposta al danno del DNA su larga scala.
Publications List
- Decitabine cytotoxicity is promoted by dCMP deaminase DCTD and mitigated by SUMO-dependent E3 ligase TOPORS
CJ Carnie, MJ Gotz, CS Palma-Chaundler, P Weickert, A Wanders, ...
The EMBO Journal, 1-27 (2024) - Transcription-coupled repair of DNA–protein cross-links depends on CSA and CSB
CJ Carnie, AC Acampora, AS Bader, C Erdenebat, S Zhao, E Bitensky, ...
Nature Cell Biology, 1-14 (2024) - Cockayne syndrome proteins CSA and CSB promote transcription-coupled repair of DNA-protein crosslinks independently of nucleotide excision repair
C Carnie, A Acampora, A Bader, V Gupta, G D'Alessandro, ...
Nature Research (2024) - USP37 prevents premature disassembly of stressed replisomes by TRAIP
OV Kochenova, G D'Alessandro, D Pilger, E Schmid, SL Richards, ...
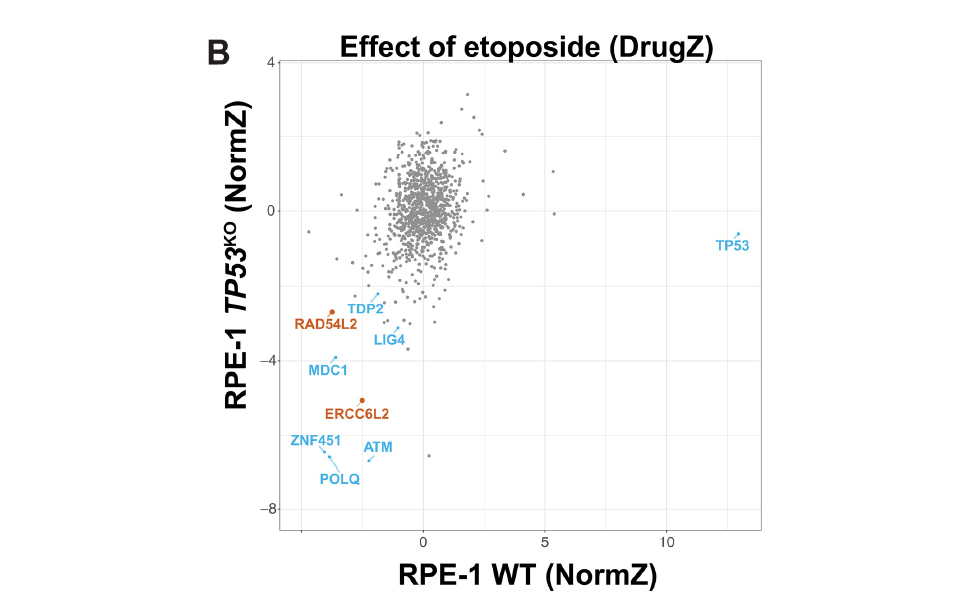
bioRxiv, 2024.09.03.611025 (2024) - RAD54L2 counters TOP2-DNA adducts to promote genome stability
G D’Alessandro, DA Morales-Juarez, SL Richards, KC Nitiss, ...
Science Advances 9 (49), eadl2108 (2023) - The dCMP deaminase DCTD and the E3 ligase TOPORS are central mediators of decitabine cytotoxicity
CJ Carnie, MJ Gotz, CS Palma-Chaundler, P Weickert, AR Wanders, ...
bioRxiv, 2023.12.21.572728 (2023) - BRCA2 controls DNA: RNA hybrid level at DSBs by mediating RNase H2 recruitment
G D’Alessandro, DR Whelan, SM Howard, V Vitelli, X Renaudin, ...
Nature Communications 9 (1), 5376 (2018) - THE ROLE OF RNA AND DNA: RNA HYBRIDS AT DNA DOUBLE-STRAND BREAKS
G D'Alessandro
Università degli Studi di Milano (2018) - Long non-coding RNA in the control of genome stability and cancer phenotypes
G D’Alessandro, FA di Fagagna
Non-coding RNA Investigation 2 (3) (2018) - A role for RNA and DNA: RNA hybrids in the modulation of DNA repair by homologous recombination
G D’Alessandro, M Adamowicz, D Whelan, SM Howard, ...
bioRxiv, 255976 (2018) - Transcription and DNA damage: holding hands or crossing swords?
G D'Alessandro, FA di Fagagna
Journal of Molecular Biology 429 (21), 3215-3229 (2017) - A damaged genome’s transcriptional landscape through multilayered expression profiling around in situ-mapped DNA double-strand breaks
F Iannelli, A Galbiati, I Capozzo, Q Nguyen, B Magnuson, F Michelini, ...
Nature Communications 8 (1), 15656 (2017) - Rearrangements of chromosome bands 15q12-q21 are secondary to HMGA2 deregulation in conventional lipoma
G Macchia, KH Nord, G D'Alessandro, J Nilsson, L Magnusson, ...
Oncology Reports 31 (2), 807-811 (2014) - NEW FUSION GENE INVOLVING EWSR1 IN ACUTE MYELOID LEUKEMIA
G Macchia, G D'Alessandro, C Lo Cunsolo, M Carella, O Palumbo, ...
HAEMATOLOGICA 97 (s2), S133-S133 (2012)